はじめに
touch-sp.hatenablog.com前回からさらに良い結果を求めてスクリプトを書き換えました。
今回使用したデータ
miniJSRT_database | 日本放射線技術学会 画像部会から「Segmentation > >Segmentation01(256×256,RGB Color:24bit)」をダウンロードさせて頂きました。学習データ50画像、テストデータ10画像です。
結果
左が前回の結果、右が今回の結果です。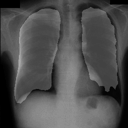
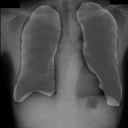
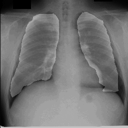

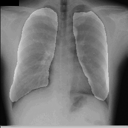
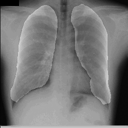
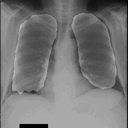
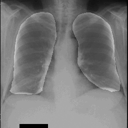
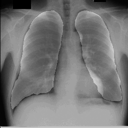
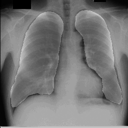
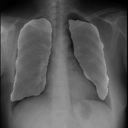
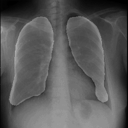
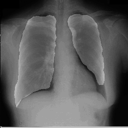
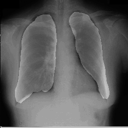
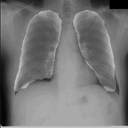
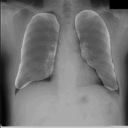
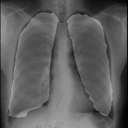
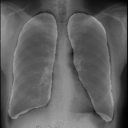
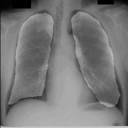
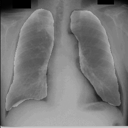
学習スクリプト
学習用画像が50枚しかないので「切り取り」と「拡大」のAugmentationを追加しました。MXNetではfixed_cropを使えば同時に実行できます。
明らかにセグメンテーションがおかしい2枚の画像は排除しました。結果的に学習に使用した画像は48枚です。
import os import glob import random import mxnet as mx from mxnet import gluon, image, autograd from mxnet.gluon.data.vision import transforms import gluoncv #hyperparameters epochs = 400 train_path = 'train_image0' ctx = mx.gpu() if mx.context.num_gpus() >0 else mx.cpu() jitter_param = 0.4 lighting_param = 0.1 input_transform = transforms.Compose([ transforms.RandomColorJitter(brightness=jitter_param, contrast=jitter_param, saturation=jitter_param), transforms.RandomLighting(lighting_param), transforms.ToTensor(), transforms.Normalize([.485, .456, .406], [.229, .224, .225]), ]) all_path = glob.glob(train_path + '/train/label/*.png') all_img = [os.path.split(x)[1] for x in all_path] def make_dataset(all_image_file): img_list = [] label_list = [] for i in all_image_file: x = random.randint(6, 10) width = int(256 * x /10) y1 = random.randint(0, 256- width) y2 = random.randint(0, 256- width) img = image.imread(train_path + '/train/org/' + i) post_image = image.fixed_crop(img, y1, y2, width, width, size=(256,256)) img_list.append(post_image) label = image.imread(train_path + '/train/label/' + i, flag=0) post_label = image.fixed_crop(label, y1, y2, width, width, size=(256,256), interp=0) post_label = post_label/255 label_list.append(mx.nd.squeeze(post_label)) train_dataset = gluon.data.dataset.ArrayDataset(img_list, label_list) return train_dataset model = gluoncv.model_zoo.FCN( root = './models', nclass = 2, backbone = 'resnet50', aux = True, ctx = ctx, pretrained_base=True, crop_size=256) lr_scheduler = gluoncv.utils.LRScheduler('poly', base_lr=0.001, nepochs=50, iters_per_epoch=len(all_img), power=0.9) criterion = gluoncv.loss.MixSoftmaxCrossEntropyLoss(aux=True) trainer = gluon.Trainer(model.collect_params(), 'sgd', {'lr_scheduler': lr_scheduler, 'wd':0.0001, 'momentum': 0.9, 'multi_precision': True}) for epoch in range(epochs): train_dataset = make_dataset(all_img) train_dataloader = gluon.data.DataLoader( train_dataset.transform_first(input_transform), batch_size=4 , shuffle=True) train_loss = 0.0 data_count = 0 for i, (data, target) in enumerate(train_dataloader): with autograd.record(): outputs = model(data.as_in_context(ctx)) losses = criterion(outputs[0], outputs[1], target.as_in_context(ctx)) losses.backward() trainer.step(data.shape[0]) data_count += data.shape[0] train_loss += mx.nd.sum(losses).asscalar() print('Epoch %d, batch %d[%d/%d], training loss %.3f'%(epoch+1, i+1, data_count, len(train_dataset), train_loss/data_count)) model.save_parameters('seg.params')